Wykresy słupkowe, zastosowanie
Procent ilości wyróżnionych w jakiś sposób danych wśród wszystkich danych pokazuje się w postaci słupków. Układ tych słupków zależy od złożoności danych i sposób wykonania wykresu powinien służyć tylko jednemu celowi: w jak najbardziej przejrzysty sposób odzwierciedlać zarówno sposób prowadzenia badań jak i uzyskane na jego podstawie wyniki. Im mniej czasu czytelnik poświęci na zrozumienie wykresu, tym lepiej.
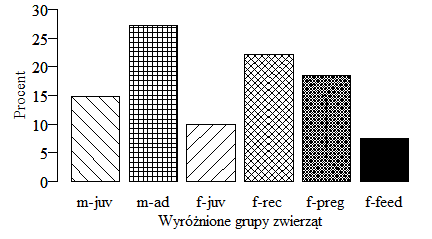
Badania drobnych ssaków z reguły polegają na rozstawieniu w środowisku pułapek żywołownych z przynętą, ich przeglądach (raz lub dwa razy dziennie), ocenie, ważeniu i znakowaniu złowionych osobników i wypuszczeniu na wolność. Obecnie znakowanie polega na zakładaniu kolczyków lub obrączek na ucho zwierzęcia (gdy celem badań jest długotrwała obserwacja populacji) lub przynajmniej wygoleniu sierści na grzbiecie zwierząt, gdy chce się odróżnić wcześniej złowione osobniki od nowych. Ocena zwierzęcia to określenie jego płci, dojrzałości i aktywności rozrodczej. Podczas takich badań złowiono 81 różnych myszy leśnych w tym 12 niedojrzałych samców, 22 dorosłych, aktywnych samców, 8 niedojrzałych samic, 18 samic dojrzałych, aktywnych i gotowych do rozrodu (tzw. receptywnych), 15 samic w ciąży i 6 karmiących. Jak najlepiej przedstawić strukturę rozrodczą populacji myszy leśnej podczas badań?
Grupa zwierząt |
Skrót |
Liczba zwierząt |
Procent |
Samce niedojrzałe |
m-juv |
12 | 14.8 |
Samce niedorosłe |
m-ad |
22 | 27.2 |
Samice niedojrzałe |
f-juv |
8 | 9.9 |
Samice receptywne |
f-rec |
18 | 22.2 |
Samice ciężarne |
f-preg |
15 | 18.5 |
Samice karmiące |
f-feed |
6 | 7.4 |
Z powyższego zestawienia widać, że skróty wyróżnionych grup zwierząt utworzono z nazw angielskich. Skrót “m” od “male”, “f” od “female”, “juv” od “juvenile”, “ad” od adult itd. Gdy praca jest przygotowywana do tłumaczenia na angielski jest to logiczne. W polskojęzycznych pracach licencjackich i magisterskich należałoby wymyślić polskie odpowiedniki tych skrótów. Skróty są używane na wykresach i w tabelach w pracy, a ich wyjaśnienie powinno znaleźć się w części “metody”. Ponieważ tym razem wykres ma się znaleźć w publikacji, a w poleceniach dla autorów znaleziono, że rozmiary wykresów powinny wynosić 4.5×2.5 cala, a używana czcionka ma mieć krój “Times New Roman”, to należy się zastosować do tych poleceń. Ponadto wydawnictwo życzy sobie wyłącznie wykresów czarno-białych, bez odcieni szarości, więc wszelkie różnice w rodzaju słupków powinny zostać wykonane za pomocą tekstury.
Polecenia związane z wykonaniem wykresu notujemy w skrypcie i ten skrypt zapamiętujemy. Sam wykres można wygenerować tuż przed posłaniem artykułu do druku, już po wszystkich poprawkach.
windows(4.5, 2.5)
windowsFonts(TimesNewRoman = windowsFont("Times New Roman"))
x=c(12,22,8,18,15,6)
par(family="TimesNewRoman", mar=c(3,3,0.5,0.5), mgp=c(1.5,0.5,0), las=1)
barplot(x/sum(x)*100,
angle=c(135,0,45,45,45,45),
density=c(10,20,10,20,50,200),
col="black", names.arg=c("m-juv","m-ad","f-juv","f-rec","f-preg","f-feed"),
xlab="Wyróżnione grupy zwierząt",
ylab="Procent",
ylim=c(0,30))
barplot(x/sum(x)*100,
angle=c(135,90,45,135,135,135),
density=c(10,20,10,20,50,200),
col="black", add=TRUE)
|
Wygenerowany wykres ma następującą postać.
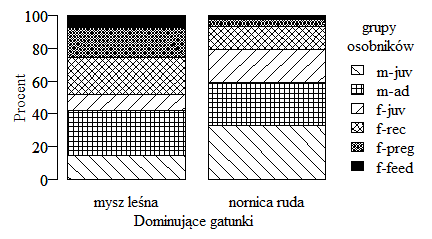
W powyższym wykresie ze względu na podpisy pod słupkami nadawanie różnych wzorów osobnikom różnej kategorii nie jest w zasadzie konieczne. Ale wiąże się to z badaniem drugiego gatunku – nornicy rudej. Złowiło się ich 107 różnych osobników, w tym 35 niedojrzałych samców, 28 dorosłych samców, 22 niedojrzałe samice, 15 samic receptywnych, 4 ciężarne i 3 karmiące. Wykres porównujący struktury płciowe obu gatunków, w najbardziej przystępny sposób pokazujący różnice w całej strukturze, wygenerujemy za pomocą poleceń:
windows(4.5,2.5)
windowsFonts(TimesNewRoman = windowsFont("Times New Roman"))
x=c(12,22,8,18,15,6)
y=c(35,28,22,15,4,3)
x1=x/sum(x)*100
y1=y/sum(y)*100
par(family="TimesNewRoman", mar=c(3,3,0.8,5), mgp=c(1.5,0.5,0), las=1, xpd=TRUE)
#
barplot(cbind(x1,y1),
angle=c(135,0,45,45,45,45),
density=c(10,20,10,20,50,200),
col="black",
axisnames=TRUE,
names.arg=c("mysz leśna","nornica ruda"),
xlab="Dominujące gatunki ",
ylab="Procent")
#
barplot(cbind(x1,y1),
angle=c(135,90,45,135,135,135),
density=c(10,20,10,20,50,200),
col="black",
axisnames=TRUE,
names.arg=c(" "," "),
add=TRUE)
#
legend(2.5, 90,
title=" \n ",
legend=c(" ", " ", " ", " ", " ", " "),
angle=c(135,0,45,45,45,45),
density=c(10,20,10,20,50,200), fill="black", box.col="white")
#
legend(2.5, 90,
title="grupy\n osobników",
legend=c("m-juv", "m-ad", "f-juv", "f-rec", "f-preg", "f-feed"),
angle=c(135,90,45,135,135,135),
density=c(10,20,10,20,50,200), fill="black", box.col="white")
|
W powyższym wykresie wyróżnienie różnych kategorii zwierząt za pomocą różnej tekstury jest konieczne. Zatem analogiczną teksturę wypada używać także na innych wykresach.
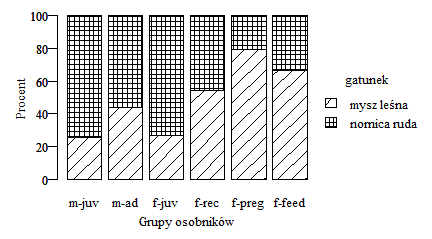
Jeszcze inny typ wykresu jest potrzebny do szczegółowego porównania obu gatunków pod względem wielkości frakcji każdej z kategorii osobników. Możliwy byłby wykres uzyskany po niewielkiej modyfikacji kodu:
windows(4.5,2.5)
windowsFonts(TimesNewRoman = windowsFont("Times New Roman"))
x=c(12,22,8,18,15,6)
y=c(35,28,22,15,4,3)
x1=x/sum(x)*100
y1=y/sum(y)*100
par(family="TimesNewRoman", mar=c(3,3,0.8,5), mgp=c(1.5,0.5,0), xpd=TRUE, las=1)
#
barplot(cbind(x1,y1), beside=TRUE,
angle=c(135,0,45,45,45,45),
density=c(10,20,10,20,50,200),
col="black",
axisnames=TRUE,
names.arg=c("mysz leśna","nornica ruda"),
xlab="Dominujące gatunki ",
ylab="Procent")
#
barplot(cbind(x1,y1), beside=TRUE,
angle=c(135,90,45,135,135,135),
density=c(10,20,10,20,50,200),
col="black",
axisnames=TRUE,
names.arg=c(" "," "),
add=TRUE)
#
legend(14.5, 30,
title=" \n ",
legend=c(" ", " ", " ", " ", " ", " "),
angle=c(135,0,45,45,45,45),
density=c(10,20,10,20,50,200), fill="black", box.col="white")
#
legend(14.5, 30,
title="grupy\n osobników",
legend=c("m-juv", "m-ad", "f-juv", "f-rec", "f-preg", "f-feed"),
angle=c(135,90,45,135,135,135),
density=c(10,20,10,20,50,200), fill="black", box.col="white")
|
Opisywana praca skoncentrowana była na zmianach struktury rozrodczej drobnych ssaków leśnych. Gdyby w większym stopniu zależało autorom na porównaniu myszy leśnej z nornicą rudą należałoby opisywane zagadnienie zobrazować inaczej. Poszczególne frakcje osobników jednego i drugiego gatunku powinny występować obok siebie. Należałoby wybrać dwie tekstury i jedną stosować konsekwentnie dla myszy leśnej, drugą dla nornicy rudej. Dwa ostatnie wykresy wyglądałyby następująco:
W pierwszym suma procentów osobników każdej grupy z obu gatunków została wyrównana do 100. Pokazuje on zatem różnice w strukturze np. młodocianych lub ciężarnych samic myszy leśnej i nornicy. W drugim suma procentów osobników z różnych grup dla każdego gatunku została wyrównana do 100 co pozwala porównywać także strukturę rozrodczą obu gatunków, ale akcentując różnice we frakcjach częściej i rzadziej łowionych osobników. Wykresy te zostały wygenerowane za pomocą programów:
windows(4.5,2.5)
windowsFonts(TimesNewRoman = windowsFont("Times New Roman"))
x=c(12,22,8,18,15,6)
y=c(35,28,22,15,4,3)
x1=x/(x+y)*100
y1=y/(x+y)*100
par(family="TimesNewRoman", mar=c(3,3,0.8,6), mgp=c(1.5,0.5,0), xpd=TRUE, las=1)
#
barplot(rbind(x1,y1),
angle=c(45,0),
density=c(10,20),
col="black",
axisnames=TRUE,
names.arg=c("m-juv", "m-ad", "f-juv", "f-rec", "f-preg", "f-feed"),
xlab="Grupy osobników",
ylab="Procent")
#
barplot(rbind(x1,y1),
angle=c(45,90),
density=c(10,20),
col="black",
axisnames=TRUE,
names.arg=c(" ", " ", " ", " ", " ", " "),
add=TRUE)
#
legend(7.3, 70,
title=" ",
legend=c(" ", " "),
angle=c(45,0),
density=c(10,20), fill="black", box.col="white")
#
legend(7.3, 70,
title="gatunek",
legend=c("mysz leśna", "nornica ruda"),
angle=c(45,90),
density=c(10,20), fill="black", box.col="white")
|
windows(4.5,2.5)
windowsFonts(TimesNewRoman = windowsFont("Times New Roman"))
x=c(12,22,8,18,15,6)
y=c(35,28,22,15,4,3)
x1=x/sum(x)*100
y1=y/sum(y)*100
par(family="TimesNewRoman", mar=c(3,3,0.8,5), mgp=c(1.5,0.5,0), xpd=TRUE, las=1)
#
barplot(rbind(x1,y1), beside=TRUE,
angle=c(45,0),
density=c(10,20),
col="black",
axisnames=TRUE,
names.arg=c("m-juv", "m-ad", "f-juv", "f-rec", "f-preg", "f-feed"),
xlab="Grupy osobników",
ylab="Procent")
#
barplot(rbind(x1,y1), beside=TRUE,
angle=c(45,90),
density=c(10,20),
col="black",
axisnames=TRUE,
names.arg=c(" ", " ", " ", " ", " ", " "),
add=TRUE)
$
legend(17, 30,
title=" ",
legend=c(" ", " "),
angle=c(45,0),
density=c(10,20), fill="black", box.col="white")
#
legend(17, 30,
title="gatunek",
legend=c("mysz leśna", "nornica ruda"),
angle=c(45,90),
density=c(10,20), fill="black", box.col="white")
|